Un equipo de científicos del Instituto Nacional de Tecnología Agropecuaria, en colaboración con la Facultad de Agronomía de la Universidad de Buenos Aires, consiguieron describir por primera vez el genoma completo de un hongo que provoca enfermedades a la planta de soja.
Se trata del Cercospora kikuchii que ocasiona el tizón morado de la hoja y la mancha púrpura de la semilla. De esta manera avanzaron en el control de ambas enfermedades provocadas a la planta.
“El conocimiento del genoma del hongo que genera la enfermedad foliar de mayor crecimiento en los últimos años en el país, permite imaginar “en un futuro cercano” el descubrimiento de moléculas fungicidas novedosas y de alto impacto para el control de estas enfermedades”, publicaron en INTA Informa, medio que difundió el hallazgo.
Con 31.1 millones de pares de bases, el genoma completo contiene 14.721 genes. El acceso a esta información aporta conocimiento para el control de las principales enfermedades que afectan a la soja.
“Es la primera vez que se describe el genoma de Cercospora kikuchii, lo que nos permite conocer el genoma del patógeno que causa la enfermedad de la mancha púrpura de la hoja”, destacó Paula Fernández, investigadora del Instituto de Biotecnología del INTA y CONICET.
Al describir el genoma “podemos revelar mecanismos esenciales de la vida del hongo, con lo cual estamos en condiciones de redescubrir productos, moléculas o agentes de control que puedan llegar a bloquear estos mecanismos que hasta hoy no eran muy conocidos, lo que nos abre una multiplicidad de opciones de estudio”, reconoció Marcelo Carmona, profesor de la Facultad de Agronomía, Universidad de Buenos Aires (UBA).
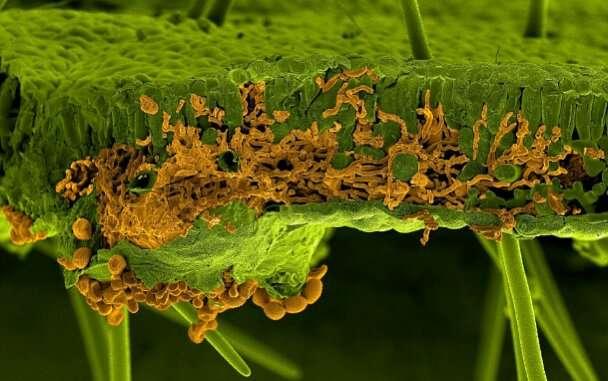
Este patógeno presenta síntomas en semillas, tallos, hojas y vainas y afecta el número y peso de los granos en el cultivo de soja. El síntoma foliar típico incluye coloración morada, bronceado a púrpura, con aspecto rugoso. Mientras que en la semilla se manifiesta un manchado morado que va desde pequeñas manchas hasta cubrirla completamente.
A partir de ahora, se tratará de conocer “los genes candidatos que afecten la infección de este patógeno en la soja, los mecanismos de posible tolerancia o resistencia a nuevos fungicidas, así como también los procesos que genera la planta para poder resistir a la infección del patógeno”, explicó Fernández.
Sumar el genoma de este hongo a la secuencia completa del genoma de la soja “descifrado hace casi 10 años”, “posibilitará identificar desde marcadores moleculares hasta genes candidatos de la infección”, reconoció la investigadora.
De acuerdo con lo publicado, las futuras investigaciones estarán dirigidas a interpretar todos los atributos con los que cuenta la planta para defenderse, así como los mecanismos del patógeno para infectar y colonizar.
Para lograr el objetivo, los investigadores de la FAUBA tuvieron la tarea de aislar el patógeno para extraerle el ADN y poder secuenciarlo, mientras que en el Instituto de Biotecnología del INTA se realizó el ensamblado mediante herramientas de bioinformática, tarea comandada por Sergio Gonzalez. La investigación contó con aportes del INTA, UBACyT y BASF.